QuantiNova LNA PCR Assay (200)
Cat. No. / ID: 249990
Eigenschaften
- Über 1,3 Millionen Assays decken die mRNA- und lncRNA-Transkripte von Mensch, Maus und Ratte in Ensembl am umfassendsten und besten ab
- Herausragende Sensitivität und Spezifität durch kurze, LNA-verstärkte Primer
- Präziser Nachweis über einen breiten Dynamikbereich ab 1 RNA-Kopie
- Zur Eliminierung unspezifischer Amplifikation optimiert
- Absolute Quantifizierung von Expressionsänderungen mittels dPCR unter Verwendung des QIAcuity Gerätes und des QIAcuity EG PCR Kit
Angaben zum Produkt
QuantiNova LNA PCR Assays setzen neue Standards in der digitalen PCR-basierten Genexpressionsanalyse. Wählen Sie aus der größten Auswahl vorgefertigter Assays für den genauen und sensitiven Nachweis jeder mRNA oder lncRNA von Mensch, Maus oder Ratte – vom allgemeinen Transkriptnachweis bis zur Differenzierung spezifischer Transkript-Isoformen. Die LNA-Verstärkung bietet uns die Möglichkeit, kürzere Primer zu entwerfen, die sich flexibler auf dem Ziel positionieren lassen. Dadurch können wir unsere Designs für den spezifischen Nachweis von Transkripten und die Differenzierung von Zielen optimieren. Darüber hinaus gewährleistet eine gründliche Designvalidierung eine optimale Leistung und robuste Erkennung. Für die digitale PCR haben wir die Assays mit den QIAcuity EG PCR Kits und dem QIAcuity Gerät optimiert und einen einfachen und schnellen absoluten Arbeitsablauf zur Quantifizierung geschaffen, der nur 2 Stunden dauert.
Beabsichtigen Sie, die QuantiNova LNA PCR Assays für die qPCR-Analyse einzusetzen? qPCR-spezifische Produkt- und Leistungsdetails finden Sie auf der entsprechenden qPCR-Katalogseite.
Sie benötigen ein Angebot für Ihr Forschungsprojekt oder möchten Ihr Projekt mit unserem Expertenteam besprechen? Nehmen Sie einfach Kontakt auf!
Leistung
Optimierte digitale PCR-Analyse mit den QIAcuity EG PCR Kits und dem QIAcuity Gerät
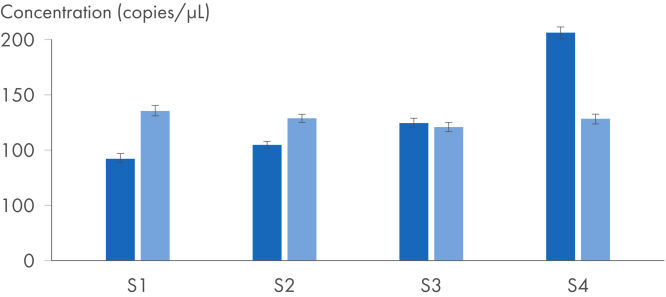
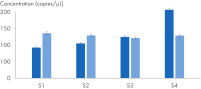
QuantiNova LNA PCR Assays dienen der hochspezifischen Zielamplifikation. Diese kann mittels zweier PCR-Methoden quantifiziert werden: qPCR mit Standard-Echtzeit-Thermocyclern und digitale PCR mit dem QIAcuity Gerät und dem QIAcuity EG PCR Kit (siehe Abbildung Nutzung der QuantiNova LNA PCR Assays bei der digitalen PCR). Nach der cDNA-Synthese mit dem QuantiTect Reverse Transcription Kit ermöglicht die digitale PCR eine hochpräzise Quantifizierung des Zielmoleküls, wobei selbst kleinste Expressionsänderungen bei geringsten Konzentrationen nachgewiesen werden. Eine Reihe von zentralen Assays wurde auf dem QIAcuity Gerät experimentell validiert.
LNA steigert PCR-Leistung
QuantiNova LNA PCR Assays wurden unter Einhaltung strenger Designkriterien und im Labor validierter Algorithmen entwickelt. Der strenge Assay-Auswahlprozess stellt sicher, dass jedes Primer-Set die zuverlässigsten und genauesten Ergebnisse bei höchster Spezifität und Effizienz liefert. Die Tm-Normierung und die LNA-Verstärkung verleihen den Primern eine höhere Bindungsaffinität als bei herkömmlichen DNA-Primern, was die Sensitivität und Spezifität des Assays deutlich erhöht.
Diese erhöhte Sensitivität ermöglicht eine herausragende Amplifikationseffizienz bis hinunter zu 1 RNA-Molekül, was den Nachweis von Zielmolekülen mit geringem Vorkommen, wie z. B. lncRNAs, aus weniger Ausgangsmaterial erleichtert. Die gesteigerte Spezifität durch die geschickte Platzierung der LNA bewirkt ein hohes Signal-Rausch-Verhältnis und ermöglicht die Unterscheidung von Sequenzen, die sich nur um ein einziges Nukleotid unterscheiden, sowie die Vermeidung von unspezifischer Amplifikation und Primer-Dimer-Bildung.
Die hohe Bindungsaffinität der LNA-Basen steigert die Flexibilität der Primerplatzierung auf dem Transkript, sodass durch intelligente Positionierung Assays für ansonsten schwer zu analysierende Ziele entwickelt werden können. Dies ermöglicht eine bessere Zielerkennung und zuverlässige Quantifizierung, selbst bei AU-reichen Zielen, Transkripten mit geringer Abundanz, Zielen mit hoher Sekundärstruktur und hochkomplexen Proben.
Vertrauen Sie den Experten für LNA-Design
Auf der Basis unserer zwanzigjährigen Erfahrung im LNA-Design waren wir in der Lage, unseren hochentwickelten LNA-Design-Algorithmus zu entwickeln und zu optimieren. Der Assay umfasst mehr als 50 verschiedene Parameter, die alle eingehend anhand strenger Leistungskriterien im Labor validiert wurden, und stellt einen erfolgreichen Zielnachweis sicher. Wir haben für jedes Transkript bis zu drei verschiedene Assay-Designs ausgewählt und alle gängigen mRNA- und lncRNA-Ziel-Assays vollständig im Nasslabor validiert. QuantiNova LNA PCR Assays funktionieren, ohne dass Sie Zeit und Geld für die Optimierung aufwenden müssen.
Prinzip
Digitale PCR auf EvaGreen-Basis
Für die absolute Quantifizierung mittels dPCR sollten Sie die QuantiNova LNA PCR Assays zusammen mit dem QIAcuity EG PCR Kit verwenden, das die Fluoreszenzdetektion auf EvaGreen-Basis nutzt. EvaGreen ist ein interkalierender Farbstoff, der an Doppelstrang-DNA bindet (ähnlich wie SYBR® Green) und bei DNA-Bindung fluoresziert. Der Einsatz von EvaGreen-basierter dPCR ist bequem und kostengünstig, da für die Amplifikation und den Nachweis des Produkts nur das Primer-Set benötigt wird. Sie bietet auch eine höhere Auflösung in der dPCR-Reaktion, die in Tausende von Partitionen der dPCR-Nanoplatte unterteilt ist, sodass die für die dPCR-Reaktion benötigte Primerkonzentration nur halb so hoch ist wie bei der qPCR.
Umfassendste und spezifischste Abdeckung
Unser proprietärer Algorithmus, der eine äußerst empfindliche, genaue und effektive mRNA- und lncRNA-Analyse sicherstellt, ermöglichte die Entwicklung von über 1,3 Millionen QuantiNova LNA PCR Assays. Die vorgefertigten Assays decken die meisten Transkripte in der Ensembl-Datenbank für menschliche, Maus- und Rattengene ab und ermöglichen PCR-basierte Genexpressionsstudien in größtmöglicher Tiefe. Die meisten Assays sind, wenn möglich, intronübergreifend und weisen nur RNA nach. Assays, die kein Intron umfassen, sind als solche gekennzeichnet, und wenn ein Exon im Ziel vorhanden ist, können unerwünschte Signale durch das QuantiTect Reverse Transcription Kit mit dem integrierten gDNA-Entfernungsschritt leicht eliminiert werden.
Wahl des richtigen Assays für Ihr Ziel
Mit den vorgefertigten QuantiNova LNA PCR Assays können Sie jede mRNA oder lncRNA von Mensch, Maus oder Ratte genau und sensitiv nachweisen, unabhängig davon, welche Analysetiefe Sie benötigen: allgemeiner Transkriptnachweis, Nachweis eines spezifischen Transkripts oder Differenzierung von Transkriptisoformen.
Die meisten menschlichen, Maus- und Rattengene haben nur ein Transkript; für diejenigen mit mehreren Transkripten haben wir nach demselben Algorithmus bis zu drei Assays pro Transkript ausgewählt. Um den unterschiedlichen Anforderungen gerecht zu werden, unterscheiden sich Design und Primerpositionierung dieser Assays. Unsere Assay-Auswahlhilfe hilft Ihnen, schnell in jeder Kategorie den besten Assay zu finden. Suchen Sie nach Assays für Ihr Ziel und wählen Sie aus den empfohlenen Assays jenen aus, der zu Ihrem Verwendungszweck passt:
- Beste Abdeckung: zur allgemeinen Transkriptdetektion.
- Bester Transkript-Assay: zum Nachweis eines spezifischen Transkripts
- Bester transkriptspezifischer Assay: zur Differenzierung spezifischer Transkript-Isoformen
Weitere Informationen finden Sie in der folgenden Tabelle.
| Generfassende Assays | Transkriptspezifische Assays | ||
| Von QIAGEN’ empfohlener Assay | Markiert als “Beste Abdeckung” | Markiert als “Bester Transkript-Assay” | Markiert als “Bester transkriptspezifischer Assay” |
| Beschreibung des Assaydesigns und der Abdeckung | Deckt die meisten biologisch relevanten Transkripte des jeweiligen Gens ab |
Hochgradig optimiert, um das interessierende Transkript spezifisch zu erkennen, wobei mehrere Isoformen des Transkripts erfasst werden |
Transkriptspezifische Spleißvariante |
| Eignung | Zum allgemeinen Transkript-Screening und zur Abklärung, ob das interessierende Gen in einer Probe exprimiert wird; zum Nachweis möglichst vieler Transkripte und Isoformen eines Gens |
Zum Nachweis eines bestimmten Transkripts | Zur Differenzierung spezifischer Isoformen eines Transkripts |
Auswertung der digitalen PCR-Daten
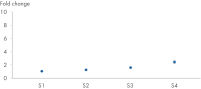
Die QIAcuity Software Suite wird zur Auswertung der dPCR-Daten verwendet und umfasst einen Genexpressionstest, der Ihre Ergebnisse als relative Expressionsänderung (Fold Change) und Fold Regulation mit publikationsreifen Abbildungen liefert.
Referenz-Gen-Assays für jede Untersuchung.
Es steht eine große Auswahl an funktionell validierten Referenz-Gen-Assays für Mensch, Maus und Ratte zur Verfügung, die eine hochwertige Datennormierung ermöglichen und zuverlässige Ergebnisse gewährleisten. Diese Assays zielen auf endogene kodierende RNA, lange nicht-kodierende RNA und kleine nukleäre RNA-Moleküle ab, die typischerweise in einer Vielzahl von Geweben konstitutiv exprimiert werden.
Normierung der mRNA/lncRNA qPCR-Ergebnisse
Die Normierung entfernt technische und biologische Abweichungen zwischen den Proben, die nicht mit den untersuchten biologischen Veränderungen zusammenhängen. Die passende Normierung ist für die korrekte Auswertung und Interpretation der Ergebnisse von Real-time PCR-Experimenten von entscheidender Bedeutung. Am häufigsten werden stabil exprimierte Referenzgene zur Normierung verwendet.
Es wird allgemein empfohlen, mehrere endogene Kontroll-Referenzgene zu testen, bevor Sie Ihre eigentliche mRNA/lncRNA-Expressionsanalyse konfigurieren. Diese Kandidaten sollten aus Genen ausgewählt werden, die voraussichtlich über die gesamte Bandbreite der untersuchten Proben hinweg stabil exprimiert werden. Dabei kann es sich um stabil exprimierte mRNA oder lncRNA handeln, die auf der Grundlage der Literatur oder bereits vorhandener Daten (z. B. NGS- oder qPCR-Panel-Screening) ausgewählt wurden. Das QuantiNova LNA PCR-System bietet validierte Referenzgen-Assays für RNA, die in der Regel stabil exprimiert wird und daher ein guter Kandidat für Referenzgene ist.
Alle Referenzgen-Kandidaten sollten für jede Untersuchung empirisch validiert werden. Eine Möglichkeit zur Normalisierung des PCR-Panels bei der Erstellung von Profilen einer großen Anzahl von mRNA/lncRNA ist die Normierung gegen den globalen Mittelwert – den Durchschnitt aller exprimierten mRNA/lncRNA. Bei Proben mit einer hohen Call-Rate (exprimierte Gene) kann dies eine gute Option sein, bei Proben mit niedrigen Call-Raten sollte man jedoch vorsichtig sein. Dies ist auch keine gute Wahl für Proben mit Veränderungen des allgemeinen Genexpressionsniveaus.
Verfahren
Zweistufige RT-dPCR
Die besten Ergebnisse werden erzielt, wenn die reverse Transkriptionsreaktion mit dem QuantiTect Reverse Transcription Kit (Kat.-Nr. 205311, 205313, 205314) durchgeführt wird. Der Einsatz des QuantiNova Reverse Transcription Kit wird nicht empfohlen. Die daraus resultierende cDNA wird dann mittels dPCR unter Verwendung der Master-Mixe des QIAcuity EG PCR Kit zusammen mit einem QuantiNova LNA PCR Assay Ihrer Wahl quantifiziert.
Wichtiger Hinweis: Die in Klammern hinter den Assay-Produktnamen angegebenen Reaktionsmengen sind für die qPCR von Bedeutung. Bei der digitalen PCR wird nur die halbe Primerkonzentration benötigt, allerdings unterscheiden sich die Reaktionsvolumina. Mit der QIAcuity Nanoplate 26K können Sie die gleiche Anzahl von Reaktionen wie angegeben konfigurieren und mit der Nanoplate 8.5K 3,3-mal so viele Reaktionen. Anweisungen zur dPCR finden Sie im Handbuch für den QIAcuity Einsatz.
Versand und Lieferung
QuantiNova LNA PCR Assays werden bei Raumtemperatur geliefert. Vorrätige Assays werden innerhalb von 1–5 Tagen geliefert. Während der Early-Access-Phase muss mit längeren Lieferzeiten gerechnet werden.
Was Sie für den Einstieg in die digitale PCR mit dem QuantiNova LNA PCR-System benötigen
Reverse Transkription: QuantiTect Reverse Transcription Kit (Kat.-Nr. 205311, 205313, 205314)
dPCR Master-Mix: QIAcuity EG PCR Kit
Assays:
- QuantiNova LNA PCR Assays
- QuantiNova LNA PCR Custom Assays
Anwendungen
QuantiNova LNA PCR Assays eignen sich hervorragend für folgende Anwendungen:
- Analyse, Profilierung und Quantifizierung der mRNA- und lncRNA-Expression
- Validierung von RNA-seq-Genexpressionsdaten
- Profilierung der Genexpression
- Signal- und Signalweganalyse
- Bestätigung des Knockdowns der Genexpression durch LNA-GapmeR bzw. siRNA
- Entwicklung von Biomarkern, einschließlich Screening, Identifizierung und Validierung von krankheitsbezogenen Biomarkern
- Überwachung phänotypischer Veränderungen im Zusammenhang mit der Genexpression
Ergänzende Daten und Abbildungen
IL-4-Genexpressionsanalyse – Nachweis kleiner Expressionsänderungen mit höchster Präzision.