✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
dPCR Microbial DNA Detection Assays
Cat. No. / ID: 250207
✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
Caractéristiques
- Détecte les espèces microbiennes, les gènes de virulence, les virus ou les gènes de résistance aux antibiotiques
- Dosages pour plus de 680 cibles
- La sélection du colorant permet de multiplexer jusqu’à 5 cibles par réaction
- Méthode de dPCR simple et rapide sur le QIAcuity
- Combiner l’ADN microbien et l’ARN viral cibles dans une réaction avec le QIAcuity OneStep Advanced Probe Kit
Détails produit
L’identification microbienne et le profilage sont intéressants dans de nombreux domaines y compris la santé humaine, l’alimentation, le test de l’alimentation et le test environnemental. L’identification microbienne détermine la présence ou l’absence d’un microbe dans un échantillon, alors que le profilage microbien détermine son expression relative dans deux conditions expérimentales ou plus, nécessitant ainsi un échantillon de référence et un normalisateur.
Nos dPCR Microbial DNA Detection Assays ciblent les gènes de résistance bactérienne, fongique, parasitaire, virale, aux antibiotiques ou les gènes du facteur de virulence. Pour les bactéries, les dosages ciblent le gène ARNr 16S et pour les champignons, ils ciblent les gènes de l’ARN ribosomal. Le portefeuille comprend plus de 680 dosages différents, dont 200 sont testés dPCR en laboratoire humide (voir les détails du dosage dans GeneGlobe).
Chaque dosage est constitué d’une paire d’amorces et d’une sonde d’hydrolyse avec un colorant fluorophore configurable. Les fluorophores disponibles sont FAM, HEX, ROX, TAMRA et Cy5, qui peuvent être mélangés et associés pour prendre en charge l’analyse de jusqu’à cinq cibles différentes dans une réaction dPCR multiplex. Nous proposons aussi des lots de dosages dPCR 5-plex testés en laboratoire humide pour analyser les cibles courantes ; voir Applications ci-dessous pour plus de détails. Pour la réaction dPCR, le dosage est combiné avec le QIAcuity Probe PCR Kit (ADN cibles) ou le QIAcuity OneStep Advanced Probe Kit (ARN cibles).
Ce kit fonctionne en conjonction avec le QIAcuity Digital PCR System et les QIAcuity Nanoplates.
Souhaitez-vous en savoir plus sur ce produit et être contacté par l’un de nos spécialistes de la dPCR ? Inscrivez-vous ici, et nous vous contacterons très prochainement.
Performances
Détection exacte et précise
Les dPCR Microbial DNA Detection Assays et le système dPCR QIAcuity permettent une quantification basée sur la PCR numérique précise. L’analyse du matériel de référence NIST a montré la concentration prévue de matrice d’entrée. Voir la figure Quantification de l’étalon de référence NIST 8376 avec l’ADNg de Shigella sonnei pour plus de détails.
Performance en multiplex
La détection des cibles microbiennes basée sur la dPCR multiplex offre une configuration flexible de petits panels en moins de réactions, ce qui préserve des échantillons précieux. Le multiplexage améliore aussi le rendement d’analyse, car il requiert moins de puits de nanoplaques et augmente le nombre d’échantillons pouvant être analysés dans chaque cycle. Nos dPCR Microbial DNA Detection Assays peuvent être combinés pour l’analyse multiplex.
Vous pouvez combiner vos propres dosages pour le multiplexage en commandant des dosages individuels avec des colorants différents (FAM, HEX, TAMRA, ROX ou Cy5). Vous pouvez aussi choisir parmi notre offre de lots de dosages 5-plex, qui ont déjà été testés en laboratoire humide en dPCR par nos scientifiques. D’autres combinaisons des plus de 685 dosages doivent être vérifiés par votre propre laboratoire.
Une comparaison entre l’analyse simplex et multiplex avec les dPCR Microbial DNA Detection Assays indique une quantification similaire et hautement précise pour toutes les cibles (voir figure Détection microbienne dans la dPCR multiplex sur QIAcuity). En plus de la quantification précise des cibles, un cycle de dPCR 5-plex montre aussi une détection hautement spécifique des différents agents pathogènes microbiens aquatiques (voir figure Détection des agents pathogènes microbiens aquatiques en 5-plex dans QIAcuity).
Principe
Les dPCR Microbial DNA Detection Assays sont conçus pour être utilisés dans la PCR numérique de nanoplaque. Chaque dosage est basé sur une amplification PCR en point final d’une région génétique spécifique de l’espèce du microbe concerné ou une région d’un gène microbien individuel. Le produit amplifié est détecté avec des sondes d’hydrolyse fluorescente spécifiques à la cible, ce qui améliore la spécificité du dosage.
Les dosages de détection des espèces bactériennes ciblent les gènes de l’ARN ribosomal – principalement le gène de l’ARN ribosomal 16S – et ont été conçus à l’aide de la base de données GreenGenes pour les séquences 16S et les séquences d’ADN de souche type déposées chez NCBI. Les dosages pour les espèces fongiques, virales et métazoaires ciblent différentes régions génétiques spécifiques à la cible, y compris des gènes d’ARN ribosomal et d’autres gènes marqueurs individuels, chacun déposé chez NCBI. Différentes bases de données ont été utilisées pour la conception des dosages des gènes de résistance aux antibiotiques (par ex. lahey.org, ARDB, etc.) et des gènes du facteur de virulence (par ex. VFDB).
Vous trouverez ici une description du principe de la réaction de dPCR sur nanoplaque.
Procédure
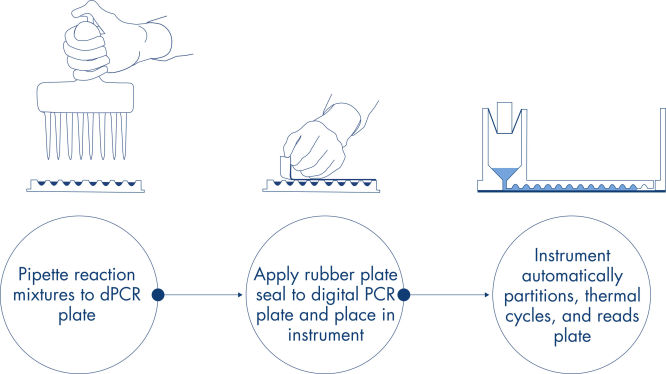
Le protocole du dPCR Microbial DNA Detection Assay est simple et peut être effectué dans tout laboratoire avec l’instrument dPCR QIAcuity. L’ADN est isolé de l’échantillon et ajouté dans le QIAcuity Probe Mastermix prêt à l’emploi et l’eau exempte d’ADN microbien (eau UCP). Ce mélange est aliquoté dans chaque puits de la pré-plaque de dPCR, qui contient des jeux pré-ajoutés d’amorces et de sondes d’hydrolyse. Les mélanges réactionnels sont transférés de la pré-plaque au puits d’une nanoplaque dPCR, qui est ensuite scellée et transférée dans l’instrument dPCR QIAcuity (voir figure Une méthode sur plaques simple et rapide). Les étapes de partitionnement, de cyclage et d’imagerie sont entièrement automatisées par l’instrument dPCR QIAcuity selon les paramètres préréglés. En fonction du protocole de cyclage, les résultats du cycle de dPCR peuvent être analysés dans la suite logicielle QIAcuity après ~2 heures (voir figure dPCR microbienne en 2 heures environ avec un temps de manipulation minimal).
Applications
Les dPCR Microbial DNA Detection Assays conviennent parfaitement à la détection des gènes de résistance aux espèces bactériennes, fongiques et virales et aux antibiotiques microbiens ou du facteur de virulence. Différents échantillons peuvent être analysés, y compris des selles, crachats, écouvillons vaginaux, eaux usées et autres.
Lots 5-plex testés par dPCR en laboratoire humide
| ID du lot | Champ d’application | Références du dosage et fluorophore* |
Cibles (ID de taxonomie NCBI) |
| WW-001 | Eaux usées 1 | DMA00278-F DMA00291-R DMA00340-H DMA00192-T DMA00344-C |
Pseudomonas aeruginosa (287) Salmonella enterica (28901) Vibrio cholerae (666) Legionella pneumophila (446) Yersinia enterocolitica (630) |
| WW-002 | Eaux usées 2 | DMA00340-H DMA00344-R DMA00199-C DMA00192-T DMA00710-F |
Vibrio cholerae (666) Yersinia enterocolitica (630) Listeria monocytogenes (1639) Legionella pneumophila (446) Coronavirus humain SARS-CoV-2 (2697049) |
| WW-003 | Eaux usées 3 | DMA00109-F DMA00192-H DMA00340-T DMA00194-R DMA00344-C |
Clostridium perfringens (1502) Legionella pneumophila (446) Vibrio cholerae (666) Leptospira alexanderi (100053) Yersinia enterocolitica (630) |
| HM-001 | Microbiome humain 1 | DMA00148-F DMA00143-T DMA00024-R DMA00003-C DMA00150-H |
Faecalibacterium prausnitzii (853) Eubacterium rectale (39491) Akkermansia muciniphila (239935) Acidaminococcus fermentans (951) Finegoldia magna (1260) |
| PB-001 | Probiotiques 1 | DMA00177-F DMA00185-T DMA00061-C DMA00137-R DMA00320-H |
Lactobacillus acidophilus (1579) Lactiplantibacillus plantarum (1590) Bifidobacterium bifidum (1681) Enterococcus faecium (1352) Streptococcus salivarius (1304) |
| RG-001 | Gènes de résistance 1 | DMA00566-F DMA00542-H DMA00576-T DMA00574-R DMA00575-C |
Gène de résistance au fluoroquinolone QnrS Groupe bêta-lactamase classe D OXA-10 Gène de résistance à la vancomycine vanB Gène tetA de la pompe d’efflux de la tétracycline Gène tetB de la pompe à efflux de tétracycline |
| RG-002 | Gènes de résistance 2 | DMA00557-T DMA00528-R DMA00548-F DMA00587-H DMA00553-C |
Gène QepA de résistance à la fluoroquinolone Groupe beta-lactamase blaVIM-1 classe B Groupe beta-lactamase OXA-48 classe D Gène sul1 de résistance au sulfonamide (43904) Groupe beta-lactamase OXA-58 classe D |
| VG-001 | Gènes de virulence 1 | DMA00614-F DMA00635-T DMA00677-H DMA00680-R DMA00597-C |
Sous-unité fimbriale mineure (fimH) Gamma-hémolysine composant B (hlgB) Toxine 1 produisant des shigatoxines sous-unité B encodée dans le prophage CP-933V Toxine produisant des shigatoxines sous-unité B ; sous-unité de liaison des récepteurs Entérotoxine du choléra accessoire (ace) |
| CP-001 | Production de cannabis 1 | DMA00278-F DMA00302-H DMA00365-R DMA00199-C |
Pseudomonas aeruginosa (287) Staphylococcus aureus (1280) Aspergillus niger (5061) Listeria monocytogenes (1639) |
| HM-002 | Microbiome humain 2 | DMA00271-F DMA00017-T DMA00049-C DMA00142-H DMA00317-R |
Prevotella oralis (28134) Actinomyces viscosus (1656) Bacteroides fragilis (817) Eubacterium infirmum (56774) Streptococcus oralis (1303) |
| RG-003 | Gènes de résistance 3 | DMA00579-F DMA00577-T DMA00573-R DMA00502-H DMA00559-C |
aac(6’)-Ib vanC oprm Groupe QnrB-1 Groupe CTX-M-1 |
| VG-002 | Gènes de virulence 2 | DMA00664-F DMA00677-H DMA00678-T DMA00642-R DMA00680-C |
ply stx1B stx2A invA stxB |
| VG-003 | Gènes de virulence 3 | DMA00688-F DMA00668-H DMA00596-T DMA00597-R DMA00611-C |
wbkA ptxA ace (E. faecalis) ace (V. cholerae) efaA |
* F : FAM, H : HEX, T : TAMRA, R : ROX ou C : Cy5
Données et illustrations utiles
Une méthode sur plaques simple et rapide