✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
QuantiTect Virus Kit (1000)
Cat. No. / ID: 211015
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- シングルアッセイだけでなくマルチプレックスアッセイでも高感度を実現
- 同一反応液中でウイルスRNA/DNAの検出が可能
- 低い陽性シグナルも明確に検出
- 迅速でユニバーサルな2ステッププロトコール
- 5x マスターミックスでサンプル添加量を増量でき感度を増大
製品詳細
パフォーマンス
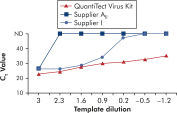
QuantiTect Virus Kitsを用いて増幅すると、希釈範囲にわたってシャープなS字曲線を示し、テンプレートが少量でも高いCT 値をとります(図 " 幅広いダイナミックレンジで明確なCT 値を測定可能")。その結果、リアルタイムPCR におけるウイルス核酸定量で正確な CT 値の決定が可能です。
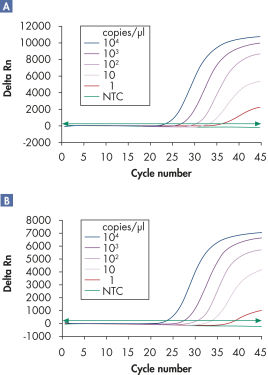
マルチプレックスアッセイにより、感度を損なうことなく複数のウイルスRNAやDNA ターゲットおよびインターナルコントロールの幅広い範囲で直線性のある検出が可能です(図 " 幅広い範囲で直線性のあるウイルスRNA 検出"および" 従来のマルチプレックスよりも低濃度でウイルスRNAを検出")。
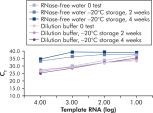
キットに同梱のQuantiTect Nucleic Acid Dilution Buffer は、希釈中および反応セットアップ中にRNAやDNAスタンダードを安定化し、チューブやピペットチップの様なプラスチック表面への核酸の吸着を防ぎます。このバッファーは、ウイルス核酸定量のためのスタンダード用検体の信頼できる希釈を可能にし、CT 値が低値から高値まで幅広い範囲で直線性のある測定を実現するとともに、スタンダード用検体の劣化のない長期保存を保証します(図 " RNAスタンダードの正確な希釈および保存")。
図参照
原理
QuantiTect Virus Kit は、シングル/マルチプレックスアッセイあるいは1ステップRT-PCRで初回実験からウイルス核酸を高感度に検出します(フローチャート" QIAGEN multiplex kits)。至適化されたマスターミックスにより、マルチプレックスPCR反応産物は、対応するシングルPCR反応産物と同じ効率と感度で増幅されます。
別々の反応ではなく同一反応内でコントロール遺伝子と標的遺伝子を増幅することで、手作業によるエラーが最小限に抑えられ、遺伝子定量の信頼性が高くなります。QuantiTect Virus Bufferに入っている画期的な合成添加剤Factor MPと、K+およびNH4+のイオン配合比により、プライマーとプローブが核酸テンプレートに効率的かつ安定してアニーリングし、高いPCR効率を実現します(図 " ユニークなPCRバッファー")。さらに、Sensiscript Reverse Transcriptaseの独自の配合により、ウイルスRNAの高感度逆転写を実現し、また、HotStarTaq Plus DNA Polymeraseにより厳密なホットスタートが可能になり、非特異的産物の形成を防ぎます。
| 成分 | 特長 | 利点 | |
|---|---|---|---|
| 5x QuantiTect Virus Master Mix | 濃縮マスターミックス | 高感度な病原体検出用に高濃縮かつ至適化済み | 高濃度マスターミックスのためテンプレート量を増やし感度を高めることが可能 |
| HotStarTaq Plus DNA Polymerase | 95℃、5分の活性化 | 室温での定量PCRのセットアップ | |
| QuantiTect Virus Buffer | NH4+イオンおよび K+イオンの配合バランス | 特異性の高いプライマーのアニーリングで信頼性の高いPCR結果 | |
| 合成添加剤Factor MP | 同一チューブで4遺伝子まで信頼できるマルチプレックス解析が可能 | ||
| 付属のキット構成 | QuantiTect Virus RT Mix | ユニークな組成のSensiscript Reverse Transcriptase | ウイルスRNAの高感度検出用に至適化済み |
| QuantiTect Nucleic Acid Dilution Buffer | 核酸スタンダードの希釈および保存に最適な独自のバッファー組成 | QuantiTect Nucleic Acid Dilution Bufferは、反応セットアップ中における標準曲線用の連続希釈RNA/DNAサンプルを安定化し、チューブやピペットチップのようなプラスチック表面への核酸の吸着を防ぐ |
図参照
操作手順
QuantiTect Virus Kitsは、配列特異的プローブを用いたウイルス核酸(RNA/DNA)および内部コントロールの高感度なリアルタイムPCR解析を実現します。逆転写反応「あり」「なし」のどちらも可能であり、標的のRNA、DNA、あるいはRNAおよびDNAの両方を検出するマルチプレックスアッセイを柔軟に設計できます。ハンドブックに記載されているプロトコールに従えば、迅速で正確な結果が得られます。
マスターミックス中にROX passive reference dyeが入ったキットまたは入っていないキットをお求めいただけます(表を参照)。
| ROX 色素 | キット | 対応するサイクラー |
|---|---|---|
| マスターミックスに添加済み | QuantiTect Virus Kit | Applied Biosystems 7500以外のApplied Biosystemsの全てのサイクラー |
| 別チューブで添付 | QuantiTect Virus +ROX Vial Kit | Applied Biosystems 7500 およびBio-Rad、Cepheid、Eppendorf、QIAGEN、Roche、Agilent、その他の会社のサイクラー |
ウイルス検出などの用途での迅速で高感度な1ステップのエンドポイントRT-PCRアプリケーション用には、QIAGEN OneStep RT-PCR Kit を推奨します。
アプリケーション
裏付けデータと数値
Unambiguous determination of CT values over a wide dynamic range.
Specifications
| Features | Specifications |
|---|---|
| ApplicationsJA | Virus detection |
| SYBR Green I or sequence-specific probes | Sequence-specific probes |
| Real-time or endpoint | Real-Time |
| Reaction type | Reverse Transcription and PCR |
| Thermal cycler | Most real-time cyclers (except capillary cyclers e.g. LightCycler® 1.x and 2.0) |
| Sample/target type | RNA and/or DNA targets |
| With or without ROX | Available with ROX in master mix and ROX as a separate vial |
| Single or multiplex | Single or multiplex |