✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
EpiTect Bisulfite Kit (48)
Cat. No. / ID: 59104
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- 迅速で信頼できる結果を実現する能率的な精製法
- 完全なシトシン変換
- ユニークなDNA保護システムにより高感度解析
- FFPE組織サンプル由来のDNA変換を含む至適化済みプロトコール
- スピンカラムおよびハイスループット用96ウェルプレートフォーマット
製品詳細
EpiTect Bisulfite Kits は非メチル化シトシンのウラシルへの完全な変換と精製を6時間以内で実現します。EpiTect 96 Bisulfite精製法の所要時間は7時間未満です。高感度なこの方法は革新的な手法によりDNA分解を防ぎ、わずか1 ngのDNAから高感度な結果が得られ、99%以上の非メチル化シトシンを変換します。独特のDNA保護テクノロジーにより、EpiTect Bisulfiteで変換したDNAはEpiTect Whole Bisulfitome Kitsを用いた全ゲノム増幅に最適です。Bisulfite変換したDNAの量が限られる場合でも、信頼性の高いDNA増幅が可能です。EpiTect Bisulfite Kitsにはスピンカラムおよび96ウェルフォーマットがあり、遠心操作あるいは吸引マニホールドを用いて処理できます。さらにEpiTect Bisulfite KitはQIAcube上で完全自動化が可能です。
パフォーマンス
迅速な結果
DNAのBisulfite処理から解析にすぐに使用できる高純度な変換DNAの分離まで、EpiTect Bisulfite Kitによる全工程(遠心法あるいは吸引法)はわずか6時間しかかかりません。EpiTect 96 Bisulfite精製法の所要時間は7時間未満です。従来のBisulfite処理法は18時間以上もかかり、非常に実験に手間取ります(表"簡単で迅速なBisulfite変換"と図" 迅速な操作手順")。EpiTect Bisulfite KitにはBisulfite変換とDNAクリーンアップを成功させるために必要な全てが含まれています。
| EpiTect精製法 | 従来の精製法 | |
|---|---|---|
| 試薬のセットアップ | 10分。分注済みですぐに使用可能 | 40分。試薬のセットアップが必要 |
| Bisulfite反応 | 5時間 | 16時間 |
| 精製 | 30~45分。EpiTect Spin Columnおよびプレート | 120分。精製、脱スルホン化、沈殿、洗浄ステップ |
完全なDNA変換
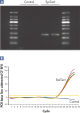
EpiTect Bisulfite Kitを用いた1回の反応で、1 ng~2 µgのDNAを同じ効率で変換できます。斬新な精製法および至適化されたバッファーにより、変換DNAは高い収量で回収できます。変換DNAをリアルタイムPCRで増幅した際に得られたCT値が非常に低いことからシトシンが高効率で変換されたことが明らかです(図" 完全なシトシン変換")。EpiTect Bisulfite KitによるDNA変換の解析結果は、非メチル化シトシンが99%以上変換されていることを示しています(図" 効率の高いシトシン変換")。この高い変換率は再現性が高く信頼できるダウンストリーム解析を実現します。
図参照
原理
微量サンプルのBisulfite変換
最小限のDNAしか得られない貴重なサンプルや限られたサンプル(例:ホルマリン固定パラフィン包埋[FFPE]組織やマイクロダイセクションで採取した組織)中のDNAメチル化パターンを決定することは特に困難です。しかし、疾病を予知する有用なバイオマーカーの同定にはこのようなサンプルタイプは特に重要です。これらのニーズに対応するため、EpiTect Bisulfite Kitにはこのようなサンプルタイプ用に至適化したプロトコールが付いています(図" FFPE組織サンプルからのDNAメチル化解析")。必要なバッファーや試薬は全てキットに入っており、最小限のマニュアルでの作業だけで実験できます。
予めDNAを分離することなくFFPEサンプル、細胞、血液、組織からDNAを直接的にBisulfite変換するためには、EpiTect Plus FFPE Bisulfite KitsおよびEpiTect Plus LyseAll Bisulfite Kitsの使用をお勧めします。
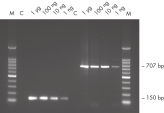
DNAの断片化の防止
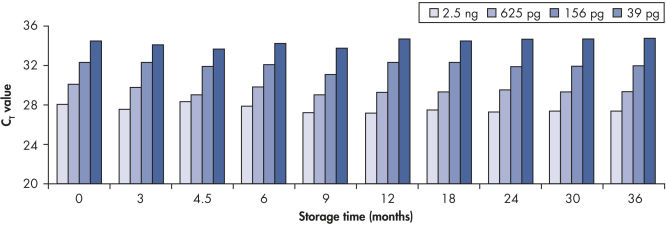
DNA Protect Bufferは、DNAの断片化を防ぐためのユニークな成分で構成され(通常DNAのBisulfite処理は高温と低いpH値で行なわれる)、効率的にDNAを一本鎖に変性し、その結果DNAを完全にシトシン変換できます(図" ユニークなDNA Protect BufferとpH指示薬システム")。断片化を防ぐことにより、長いPCR増幅産物が得られ解析できます(図" 簡単で迅速なBisulfite変換")。さらにDNA Protect BufferにはpH指示薬が含まれているので、シトシン変換のための正確なpHを確認できます。この方法に組み込まれた効率的なDNAクリーンアップにより、DNA品質に影響を及ぼすことなく、変換DNAを少なくとも12ヶ月間保存できます(図" DNAの長期保存")。
図参照
操作手順
EpiTect精製法はQIAGENキットの特長である結合、洗浄、溶出システムを利用した簡単な操作工程で構成されている(フローチャート" 変換手順")。加熱変性とsodium bisulfite処理によるDNA変換の後、DNAをEpiTect Spin Columnあるいはプレートにアプライし、残留している全てのsodium bisulfiteを除去するため至適化されたバッファーおよび一般的なマイクロ遠心機あるいは吸引マニホールドを用いて洗浄し、その後DNAを溶出する。溶出されたDNAはメチル化に特異的なPCR(Methylation-specific PCR)、リアルタイムPCR解析、Bisulfiteシークエンシング、COBRA、パイロシークエンシングを含む全てのダウンストリームアプリケーションに使用できます。
エピジェネティクス情報を得ることは生物学や医学研究における多くの分野(特に、腫瘍学や幹細胞研究、および発生生物学)にとって最も重要です。しかし、DNAメチル化における変化の解析はサンプル(特に非常に限られた量のサンプル)から再現性のあるデータを得るための標準化された方法がないために大変な仕事です。QIAGENが新しく紹介するEpiTectソリューションにより、DNAサンプル採取、安定化、精製からBisulfite処理、リアルタイム/エンドポイントPCRメチル化解析またはシークエンシングまで、標準化されたプレアナリティカル用製品および解析用製品を提供いたします。(図" エピジェネティクスにおける標準化されたワークフロー")。
図参照
アプリケーション
EpiTect Bisulfite Kitを用いて変換、精製したDNAは、以下のような幅広いダウンストリームアプリケーションでの使用に最適です。
- メチル化に特異的なPCR(Methylation-specific PCR)、リアルタイムPCR
- COBRA
- シークエンシング/パイロシークエンシング/NGS
- HRM(High Resolution Melt)
- メチル化解析用アレイ
裏付けデータと数値
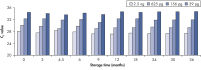
Long-term DNA storage.
Bisulfite converted DNA generated with EpiTect Kits was stored at –20°C for up to 36 months, and amplified at several time intervals. The resulting threshold cycle values at all time points for various DNA amounts were comparable to those obtained with freshly converted DNA (time point 0).
Specifications
| Features | Specifications |
|---|---|
| ApplicationsJA | Multiplex PCR, real-time PCR, sequencing |
| Processing | Manual |
| Sample types | Blood, (FFPE) tissue, DNA samples |
| Conversion efficiency | 99.4–99.8% |
| Format | Spin column and 96-well plate |
| Time per run or per prep | <6 hours (1-48 samples), ~ 7 hours (48-96 samples) |
| Elution volume | Varies |
| Sample amount | 1–2 µg |
| Yield | Depends on input amount of purified genomic DNA used for conversion |