✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
QIAamp DNA Microbiome Kit (50)
Cat. No. / ID: 51704
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- スワブおよび体液から細菌由来のDNA を分離
- 宿主由来のDNA を効果的に除去
- 最適化された細胞の機械的破砕および化学的な溶解
- ウルトラクリーンなカラムでコンタミリスクを最小限化
製品詳細
QIAamp DNA Microbiome Kitは、スワブや体液からマイクロバイオーム由来のDNAを精製・濃縮するための専用製品です。精製工程における効果的な宿主DNA の除去は、次世代シークエンシング解析において細菌DNA のカバレッジを最大限にし、高感度な16S rDNA をベースにしたマイクロバイオーム解析やメタゲノムのショットガンシークエンスを実現します。
パフォーマンス
スワブや体液には細菌細胞だけではなく、宿主であるヒトや動物細胞由来のDNA も含まれています。メタゲノム研究では、サンプルからゲノムDNA を分離して解析を行ないますが、宿主であるヒトや動物由来のDNA が細菌DNA よりも多く含まれており、これらのDNA はマイクロバイオーム解析を妨害することがあります。実際にヒトマイクロバイオーム計画(Human Microbiome Project)で実施された異なるサンプル種の全メタゲノム・ショットガンシークエンシングにおける重要な発見の一つは、シークエンスで得たリードの99%までがヒトゲノムDNA に相当しており、細菌由来のDNA は多くても1%しかありませんでした。16S rDNA シークエンシングと比較して全メタゲノム・ショットガンシークエンシングは、病原性因子、抗生物質耐性の存在、あるいは代謝ネットワークのようなマイクロバイオーム研究に貴重な洞察を付加できます。このように、細菌由来のリードのカバレッジを最大化することにより解析能力が大幅に強化されます。
宿主のDNA 除去は、シークエンシング実験における細菌由来リードのカバレッジを増大します。QIAamp DNA Microbiome Kit は、宿主細胞だけを溶解し、続いて宿主由来のDNA を酵素分解することで宿主のDNAを効率的に除去します。その後、機械的破砕および化学的な溶解を組み合わせた処理により、インタクトな細菌細胞を効率的に溶解し、遊離した細菌由来のDNA を実績のあるQIAamp ケミストリーとコンタミ除去されたQIAamp UCP スピンカラムを用いて精製します(図 宿主由来のDNA 除去を組み込んだ精製法)。
全メタゲノム・ショットガンシークエンシング実験における細菌由来のDNA 成分が増大
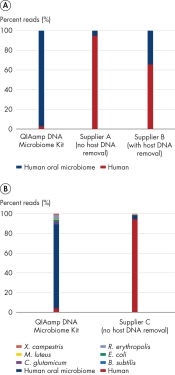
全メタゲノムのショットガンシークエンシング実験において宿主ゲノムに由来するリードの割合が高い場合、高性能なバイオインフォマティックス・ツールを利用した場合でさえ、細菌データベースの適切なアセンブリに時間も人手もかかってしまいます。QIAamp DNA Microbiome Kit は、全メタゲノムのショットガンシークエンシング用にサンプル中の宿主DNA を除去した細菌由来のDNA が濃縮されたサンプルを提供します。ヒト口腔スワブからQIAampDNA Microbiome Kit、あるいは他の2 社製品を用いてDNA 調製し、分離したDNA を用いて全メタゲノムのショットガンシークエンシングを実施したリード結果を比較しました。 ヒトDNAに由来するリードの割合は、QIAamp DNA Microbiome Kit を用いて調製したサンプルのシークエンシングにおいて5%以下であり、他社の宿主由来のDNA 除去がないキットで90%以上、DNA 除去を含むキットで35%という結果と比較して大幅に抑えられました。 次の実験では、サンプルに既知の培養細菌を添加しました。宿主由来のDNA 除去がない製品に比べて、本キットでは添加した細菌のDNA がより正確な組成比で回収できました(図 効果的な宿主由来のDNA 除去による全メタゲノム・ショットガンシークエンシング結果の向上)。
16S rDNA シークエンシングにおける増幅改善
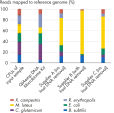
16S rDNA シークエンシングは細菌群における相対的な構成パターンを同定するために一般的に使用されています。3種類の他社精製キットと比較した場合、QIAamp DNA Microbiome Kitは、口腔スワブから精製したDNA におけるV4 領域の最も効果的な増幅を実現します(図 16S rDNA の効率的な増幅およびシークエンシング)。QIAamp DNA Microbiome Kit を用いて精製した2 サンプルから増幅したV4 領域のシークエンシング結果は、ヒト口腔マイクロバイオームで予測した細菌の構成パターンを示しました。
至適化済みの細胞溶解でサンプル調製によるバイアスを最小限化細胞壁の形態により、細菌は異なる溶解法に対して様々な溶解反応を示します。例えば、脂質や多糖類に富む厚い細胞壁を有する細菌は、酵素による溶解法で調製したサンプル中では実際より少ない構成内容として提示される傾向があります。その結果、いずれか一種類の溶解法を用いた場合には、マイクロバイオームの正確な構成内容および相対的な組成比においてバイアスを引き起こすことがあります。QIAamp DNA Microbiome Kit は、サンプル調製により誘導されるバイアスを最小限にするために最適化された機械的な破砕法と化学的な溶解法を組み合わせて用いています。さらに、QIAamp DNA Microbiome Kit に入っているUCP(Ultra Clean Production)スピンカラムは、コンタミリスクを最小限にするために独自のクリーニング工程で製造されています。
サンプル調製によるバイアスを調べるため、既知のコロニー形成単位(CFU;colony forming units)の細菌6 種類を含む細菌群モデルを培養細胞から作製しました。そのマイクロバイオーム・モデルのサンプルをQIAamp DNA Microbiome Kit、あるいは代わりのキットを用いてシークエンシング用に調製しました。QIAamp DNA Microbiome Kit を用いて調製したサンプルは、使用した他のすべての方法と比べてマイクロバイオーム・モデルの組成比をより正確に示しました(図 サンプル調製によるバイアスは最小限)。
図参照
原理
操作手順
図参照
アプリケーション
QIAamp DNA Microbiome Kitは混合サンプルからマイクロバイオーム由来のDNAを精製および濃縮し、以下のような高感度なダウンストリーム・アプリケーションに使用できます:
- 全ゲノムシークエンシング解析
- 高感度な16S rDNAによるマイクロバイオーム解析
- メタゲノミックなショットガンシークエンシング研究
裏付けデータと数値
Effective host DNA removal enhances whole metagenome shotgun sequencing results.