✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
QIAseq miRNA Library Kit (12)
Cat. No. / ID: 331502
For 12 sequencing prep reactions: 3’ ligation, 5’ ligation, reverse-transcription, cDNA cleanup, library amplification and library cleanup reagents; quality control primers
KitIndex
Library Kit
miRNA Index Kit Version 1
miRNA Index Kit UDI Version 2
Reactions
12
96
QIAseq miRNA Library Kitは分子生物学的アプリケーション用であり、疾病の診断、予防、あるいは治療に使用することはできません。
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- わずか1 ngの全RNAからゲルフリーmiRNAシーケンシングライブラリー調製
- アダプターダイマーおよび不要RNA種の除去による、最高のフィデリティと高効率データ
- ビーズベースでのアダプターダイマーおよび不要RNA種除去
- 個々のmiRNA分子の定量を可能にするIntegrated Unique Molecular Indices(UMI)
- GeneGlobe Data Analysis Centerを使用した一次リードマッピングおよび差次的発現解析
製品詳細
最適化された反応は、反応バイアスを最小限に抑え、アダプターダイマーを排除しながら、確実なmiRNA特異的ライブラリー調製を可能にします。 Unique Molecular Indices(UMI)は反応初期段階で各miRNAをタグ付けし、PCRおよびシーケンシングバイアスを排除します。 GeneGlobeを介した無料の統合されたデータ解析により、miRNAマッピングとUMI解析、および差次的発現解析が可能です。
パフォーマンス
QIAseq miRNAシークエンシングにより、改善されたワークフローにより優れたデータ生成が可能です。 標準的なQIAseq miRNA法は、ゲル精製・切り出しおよび溶出を必要とせず、サンプル損失を防ぎ、処理時間を短縮します。
miRNAシーケンシングの大きな問題は、アダプターダイマーの混入があることです。 QIAseq miRNA Library Kitは完全に最適化されたライブラリープロセスを採用し、非常に低いtotal RNA量でもアダプターダイマーを実質的に排除します( アダプターダイマー(AD)と汚染RNAがmiRNAseq実験のリードを妨害を参照)。 さらに反応は、確実なmiRNA作製に焦点を絞り、バイアスとバックグラウンドの混入を排除します。 これらの利点により、QIAseq miRNA Library Kitは、シーケンスに利用可能なmiRNAの収量を最大化することができます。
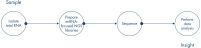
QIAseq miRNA Library Kitは、潜在的なバイオマーカーとしての循環型miRNAに対応するために、低入力レベルにまでmiRNAをマッピングできるように最適化されています。 さらに、逆転写プロセス中にUnique Molecular Indices(UMI)を導入し、NGSによる成熟miRNAの不均一かつ正確なmiRNome全体定量を可能にします。 QIAseq miRNAは、次世代シークエンシングを使用して、差次的発現解析と新規miRNA発見のための比類のないSample to Insightソリューションを提供します( QIAseq miRNAワークフローを参照)。
miRNAシーケンシングの大きな問題は、アダプターダイマーの混入があることです。 QIAseq miRNA Library Kitは完全に最適化されたライブラリープロセスを採用し、非常に低いtotal RNA量でもアダプターダイマーを実質的に排除します( アダプターダイマー(AD)と汚染RNAがmiRNAseq実験のリードを妨害を参照)。 さらに反応は、確実なmiRNA作製に焦点を絞り、バイアスとバックグラウンドの混入を排除します。 これらの利点により、QIAseq miRNA Library Kitは、シーケンスに利用可能なmiRNAの収量を最大化することができます。
QIAseq miRNA Library Kitは、潜在的なバイオマーカーとしての循環型miRNAに対応するために、低入力レベルにまでmiRNAをマッピングできるように最適化されています。 さらに、逆転写プロセス中にUnique Molecular Indices(UMI)を導入し、NGSによる成熟miRNAの不均一かつ正確なmiRNome全体定量を可能にします。 QIAseq miRNAは、次世代シークエンシングを使用して、差次的発現解析と新規miRNA発見のための比類のないSample to Insightソリューションを提供します( QIAseq miRNAワークフローを参照)。
図参照
原理
成熟miRNAは、転写後遺伝子調節を仲介する、天然の22ヌクレオチドのnon-coding RNAです。 miRNAの改変は、発生、分化、シグナル伝達、感染、老化および疾患における遺伝子発現の変化と相関します。血清、血漿、脳脊髄液(CSF)および尿を含むすべての生体液中で発現されるmiRNAとして、循環miRNAと正常および疾患生物学を関連付ける証拠が継続的に報告されています。特に癌においては、多くの研究とレビューが、様々なmiRNAの存在と癌細胞増殖、アポトーシスに対する抵抗性、侵襲性および分化を関連づけています。
miRNA発現の定量は、次世代シークエンシング(NGS)およびリアルタイムPCR(qPCR)を含む様々な技術で行うことができます。 NGSは新規miRNA発見のためのデフォルトツールですが、市販のライブラリー調製キットは煩雑でバイアスを導入します。 結果として、これまでmiRNA発現定量にはqPCRが用いられていました。 QIAseq miRNAは、新世代のsmall RNAシークエンシング製品で、他のシーケンシングキットにはない特長を含んでいます。 QIAseq miRNA Library Kitを使用することで、UMIによる単一分子の定量とNGSの能力を組み合わせて、最も代表的な発現データを得ることができるようになりました。
miRNA発現の定量は、次世代シークエンシング(NGS)およびリアルタイムPCR(qPCR)を含む様々な技術で行うことができます。 NGSは新規miRNA発見のためのデフォルトツールですが、市販のライブラリー調製キットは煩雑でバイアスを導入します。 結果として、これまでmiRNA発現定量にはqPCRが用いられていました。 QIAseq miRNAは、新世代のsmall RNAシークエンシング製品で、他のシーケンシングキットにはない特長を含んでいます。 QIAseq miRNA Library Kitを使用することで、UMIによる単一分子の定量とNGSの能力を組み合わせて、最も代表的な発現データを得ることができるようになりました。
操作手順
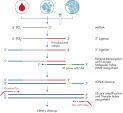
QIAGENは、試料分離からデータ分析および解釈まで、真のSample to Insightワークフローを提供します。 miRNeasyキットを使用して、生体液(血清、血漿、CSFおよび尿など)、細胞、新鮮/凍結組織またはFFPE組織から全RNAを最初に抽出します。 そこから、QIAseq miRNA Library Kitを使用してmiRNAシークエンシングライブラリーを調製します(図を参照)。
バイアスの無い反応では、アダプターをmiRNAの3 '末端および5'末端に連続して結合します。 続いて、UMIを導入したcDNA合成、cDNAクリーンアップ、ライブラリー増幅およびライブラリークリーンアップが行われます。 独自の方法により、修飾オリゴヌクレオチドを用いて、シークエンシングライブラリー中のアダプターダイマーを排除し、シークエンシング時に観察される主要な汚染物質を効果的に除去します。 ビーズベースのクリーンアップは、不要なバックグラウンドノイズを排除します。 UMIは、増幅またはシークエンシングアーティファクトではなく、サンプルが特異的にされていることを保証します。 サンプルからシーケンサーに移るまでに、わずか8時間しかかかりません。 最大48サンプルをマルチプレックス化することができます。
シーケンシングの後、 ".fastq"または ".fastq.gz"ファイルをGeneGlobe Data Analysis Centerに直接アップロードして、一次マッピングと分子タグカウントを行うことができます。 ヒト、マウス、ラットなどのよく特徴付けられた種については、種特異的miRBaseおよびゲノムデータベースにマップされます。 特徴づけられていない種または新規種については、miRBaseデータベース全体にマッピングされます。 次いで、分子タグ数計測および得られたデータの視覚化をとおして差次的発現解析を行います。
バイアスの無い反応では、アダプターをmiRNAの3 '末端および5'末端に連続して結合します。 続いて、UMIを導入したcDNA合成、cDNAクリーンアップ、ライブラリー増幅およびライブラリークリーンアップが行われます。 独自の方法により、修飾オリゴヌクレオチドを用いて、シークエンシングライブラリー中のアダプターダイマーを排除し、シークエンシング時に観察される主要な汚染物質を効果的に除去します。 ビーズベースのクリーンアップは、不要なバックグラウンドノイズを排除します。 UMIは、増幅またはシークエンシングアーティファクトではなく、サンプルが特異的にされていることを保証します。 サンプルからシーケンサーに移るまでに、わずか8時間しかかかりません。 最大48サンプルをマルチプレックス化することができます。
シーケンシングの後、 ".fastq"または ".fastq.gz"ファイルをGeneGlobe Data Analysis Centerに直接アップロードして、一次マッピングと分子タグカウントを行うことができます。 ヒト、マウス、ラットなどのよく特徴付けられた種については、種特異的miRBaseおよびゲノムデータベースにマップされます。 特徴づけられていない種または新規種については、miRBaseデータベース全体にマッピングされます。 次いで、分子タグ数計測および得られたデータの視覚化をとおして差次的発現解析を行います。
アプリケーション
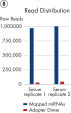
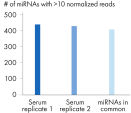
QIAseq miRNA Library Kitは、total RNAおよびmiRNAを含む、血清、血漿、CSF、尿などの生体液、細胞、新鮮/凍結組織、FFPE組織に適合しています。 QIAseq miRNAを使用して、次の循環miRNAバイオマーカーを特性化することができます。 FFPE組織に閉じ込められたmiRNAシグネチャーを明らかにすることができます。 新しい種を完全に特性化することができます。 QIAseq miRNA Library Kitは、血清などの生体液からの収量を高めるように設計されています。 miRNA検出の図では、QIAseq miRNA Library Kitによる血清サンプルからのmiRNAの確実な検出を示しています。 次いで、マッピングされたリードを、血清サンプル中のアダプターダイマーと比較しました。 QIAseq miRNAは、限られたサンプルでもmiRNAの優れたマッピングを示しています( 血清サンプル中のリード分布を参照)。 QIAseq miRNA Library Kitを使用して、任意の種由来の全RNAサンプルから、既知または新規のmiRNAの差次的な発現を決定することができます。
QIAseq miRNAは、数百サンプルを処理する大規模プロジェクトから、標的miRNAのグループに焦点を当てた小さなパイロットまで、発見と発現を強化する究極のツールです。 QIAseq miRNAは、次世代シークエンシングを用いた差次的発現解析と新規miRNAの発見のための比類のないSample to Insightソリューションを提供します。
QIAseq miRNAは、数百サンプルを処理する大規模プロジェクトから、標的miRNAのグループに焦点を当てた小さなパイロットまで、発見と発現を強化する究極のツールです。 QIAseq miRNAは、次世代シークエンシングを用いた差次的発現解析と新規miRNAの発見のための比類のないSample to Insightソリューションを提供します。
図参照
裏付けデータと数値
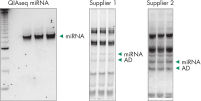
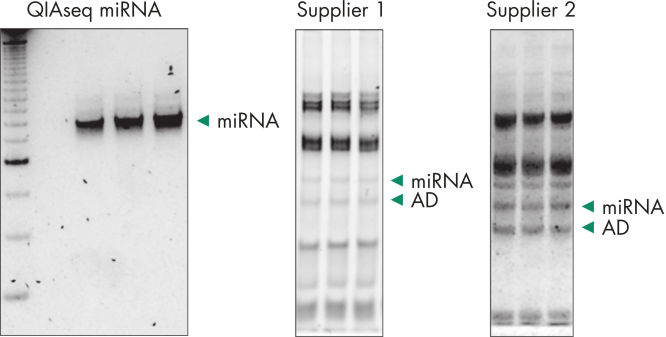
Adapter dimers (AD) and contaminating RNAs steal your reads during miRNA sequencing experiments
While gels can be used to eliminate adapter dimers and contaminating RNAs, there is still a possibility for high prevalence in sequencing reads even after gel excision. On the above left, QIAseq miRNA shows a robust miRNA library with no adapter dimers or contaminating RNA after the basic protocol that includes a bead-based purification. Compared to libraries generated with competitor kits (prior to a required tedious gel excision), the QIAseq-derived miRNA library is much more robust and devoid of adapter dimers and contaminating RNAs.
リソース
クイックスタートプロトコール (4)
機器テクニカル資料 (11)
パンフレット (2)
キットハンドブック (4)
サイエンティフィック・ポスター (1)
MSDS (1)
Supplementary Protocols (2)
Safety Data Sheets (1)
Certificates of Analysis (1)
FAQ
What is the recommended read length for libraries prepared using the QIAseq miRNA Library Kit?
Can you perform qPCR analysis of miRNAs from libraries prepared with the QIAseq miRNA Library Kit?
Total RNA from what sample types are compatible with the QIAseq miRNA Library Kit?
What method is used to eliminate adapter dimers from the QIAseq miRNA library?
Are any components of the QIAseq miRNA Library Kit interchangeable with components from other QIAGEN kits?
Should total RNA or small enriched RNA be used as the starting material for the QIAseq miRNA Library Kit?
What is the sequence of the QIAseq miRNA NGS 5’ Adapter?
Are the QIAseq miRNA NGS 5’ Adapter and QIAseq miRNA NGS RT Initiator compatible with the QIAseq miRNA UDI kits?
What is the maximum number of available sample indexes?
What can be used to validate results obtained with the QIAseq miRNA Library Kit?
What is the acceptable number of reads per sample per miRNA sequencing run?
How do Unique Molecular Indexes (UMIs) improve quantification?
What are recommended stopping points during the QIAseq miRNA Library Kit procedure?
Does a QIAseq miRNA library include hY4 Y RNA?
Can I still perform sequencing if my miRNA sequencing library concentrations are too low to obtain a 4 nM library?
When multiplexing samples, what sample indexes should be combined?
What are common sized libraries observed on a TapeStation, Bioanalyzer, or similar instruments?
Can miRNA sequencing libraries be prepared from total RNA isolated from heparinized plasma samples?
What is the sequence of the QIAseq miRNA NGS 3’ Adapter?
What is the sequence of the UDI 5' Adapter?