AllStars Negative Control siRNA
RNAi実験でのネガティブコントロール
RNAi実験でのネガティブコントロール
AllStars Negative Control siRNAの性能は表に記載の実験により検証しました。
| テストの種類 | テストの対象 | 目的 | AllStars Negative Control siRNA での結果 |
|---|---|---|---|
| ゲノムワイド解析 | Affymetrix GeneChip アレイ | 遺伝子発現の非特異的な制御 | 制御された遺伝子数は非常に少ない |
| 細胞ベースのアッセイ | 生存細胞の核染色 | 核の大きさ | 正常 |
| 細胞ベースのアッセイ | 細胞数 | 増殖率 | 変化なし |
| 細胞ベースのアッセイ | ヌクレオチドの取り込み | DNA 合成率 | 変化なし |
| 細胞ベースのアッセイ | 生存細胞の色素排除 | 細胞毒性の効果 | 変化なし |
| 細胞ベースのアッセイ | DNA 染色 | 細胞周期の配分 | 正常 |
| RISC への取り込み解析 (HeLa および MCF-7 細胞) | レポーターコンストラクトのトランスフェクション | siRNA が RISC に取り込まれるかどうかを調査 (有効なコントロールsiRNA はRISC に入り込む) | RISC に取り込まれた |
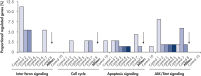
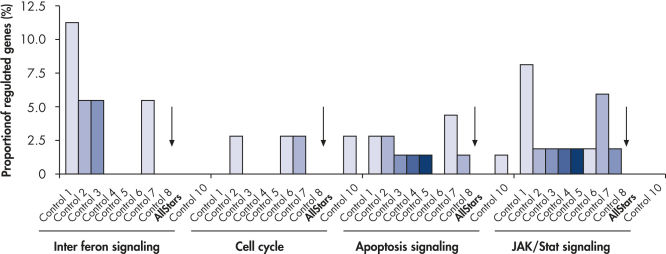
複数のネガティブコントロールsiRNAのトランスフェクション後、遺伝子発現への非特異的な影響のレベルをテストするためにゲノムワイドな解析を行ないました。HiPerFect Transfection Reagentを用いて異なる由来の複数のネガティブコントロールsiRNAをMCF-7、K562、初代HUVEC細胞にトランスフェクトしました。これらは、Nonsilencing siRNA(哺乳動物遺伝子と相同性がない)、スクランブルsiRNA(遺伝子特異的なsiRNAと同じ塩基組成を持つが配列が異なる)、人工的なレポーター遺伝子を標的にしたsiRNAが含まれています。続いて、Affymetrix GeneChipアレイを用いてヒト全ゲノムの発現プロファイリングを行ないました。AllStars Negative Control siRNAは非特異的に制御される遺伝子数が常に最も少ないことから、ネガティブコントロールに最適です。一方、他のネガティブコントロールsiRNAは重要な細胞内パスウェイに関与する多くの遺伝子を非特異的に抑制しました(図“ 発現への非特異的な影響が低い”)。
生存細胞の核染色を用いて核サイズを測定しました。サイズ変化は細胞周期障害や増殖阻害を示唆している可能性があります。様々なタイプのネガティブコントロールsiRNAを用いてテストを行ないました。AllStars Negative Control siRNAは、テストしたコントロールsiRNAの中で最高の結果を提供しました。AllStars Negative Control siRNAのトランスフェクションは、トランスフェクトしていない細胞と比較して、核の大きさに変更はありませんでした。一方、他のnegative control siRNA(Control 1)では核が拡大しました(図“ 核サイズの表現型は影響を受けない”)。
細胞数は、細胞が正常に増殖していたかどうかを判断するために様々なネガティブコントロールsiRNAのトランスフェクション後に評価しました。未処理の細胞とAllStars Negative Control siRNAをトランスフェクトした細胞では細胞数の違いはほとんど観察されませんでした。一方、その他のテストしたネガティブコントロールsiRNAのトランスフェクション後では細胞数は顕著に減少しており、Control 1のようにこれらのsiRNAが細胞増殖を抑制することを示唆しています(図“ 細胞数に影響を与えないsiRNA”)。
トランスフェクトしていないHCT-116細胞と様々なネガティブコントロールsiRNAでトランスフェクトしたHCT-116細胞におけるDNA合成速度を決定するために、ヌクレオチドの取り込みを測定しました。ヌクレオチド取り込みはブロモデオキシウリジン(BrdU)取り込みを調べることにより測定しました。これはチミジンの塩基アナログで、DNA複製時のチミジンの代替として、新たに合成されたDNAに組み込まれます。 DNA合成率の変化は、細胞増殖または細胞周期の変化を示しています。 AllStars Negative Control siRNAをトランスフェクトした細胞のBrdU取り込み率は、トランスフェクトしていない細胞のそれに非常に類似していました。 しかし、テストした他のネガティブコントロールsiRNA(Control 1)では、DNA合成レベルの低いプロファイル変化が得られ、このsiRNAが細胞増殖や細胞周期に影響を与えることを示唆しています(図“ 通常のDNA合成における表現型”)。
生細胞色素排除は様々なネガティブコントロールsiRNAの潜在的な細胞毒性効果を測定するために使用されていました。 AllStars Negative Control siRNAでトランスフェクトした細胞とトランスフェクトしていない細胞は、生細胞と死細胞の数で類似した結果が得られました。対照的に、他のネガティブコントロールsiRNAでは細胞毒性の増加が観察されました(図“ 細胞毒性効果の増加なし”)。
細胞周期分布を測定するために細胞固定の後にDNA染色を用いました(細胞周期G1/G0、S、G2)。AllStars Negative Control siRNAのトランスフェクション後、各周期での細胞の比率はトランスフェクトされていない細胞のものと類似しています(図“ 正常の細胞周期分布”)。この結果はAllStars Negative Control siRNAが細胞周期に悪影響を及ぼさないことを証明しています。
正確なネガティブコントロールRNAi実験のために、ネガティブコントロールsiRNAはRISC(RNA-Induced Silencing Complex)に組み込まれるべきです。 これは、コントロールsiRNAが遺伝子特異的なsiRNAと同様の生物学的プロセスを通過し、遺伝子特異的なsiRNAからのデータをネガティブコントロールsiRNAからのデータと比較でき、ターゲット遺伝子のノックダウンに起因するという結果を決定できることを意味します。
実験は以下のように行ないました。
レポーター・コンストラクトと非相補的siRNAのコトランスフェクションは、蛍光タンパク質とHisタグの強い発現をもたらしました。 コンストラクトをAllStars Negative Control siRNAとコトランスフェクトした場合には、siRNAは相補配列からの発現をノックダウンし、その結果、蛍光レポーター遺伝子、Hisタグ、およびsiRNAターゲット配列をコードする全mRNA転写物の分解が生じました。mRNA分解は融合タンパク質のノックダウンを引き起こします(図“ AllStars Negative Control siRNAのRISCへの取り込み”および“ AllStars Negative Control siRNAのRISCへの取り込みを示すウエスタンブロット解析”)。クローニング実験によりAllStars Negative Control siRNAはRISCに取り込まれることが確認されています。
 Nuclear size phenotype unaffected.
Nuclear size phenotype unaffected.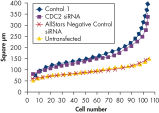 Cell number unaffected.
Cell number unaffected.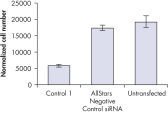 Normal DNA synthesis phenotype.
Normal DNA synthesis phenotype. No increase in cytotoxic effects.
No increase in cytotoxic effects. Normal cell-cycle distribution.
Normal cell-cycle distribution. Reporter construct for RISC-incorporation experiment
Reporter construct for RISC-incorporation experiment AllStars Negative Control siRNA is incorporated into RISC.
AllStars Negative Control siRNA is incorporated into RISC.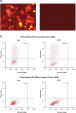 Western analysis shows AllStars Negative Control siRNA enters RISC.
Western analysis shows AllStars Negative Control siRNA enters RISC.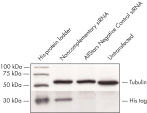
全てのRNAi実験でネガティブコントロールsiRNAをトランスフェクトすることが重要です。ネガティブコントロールでの結果を未処理の細胞での結果と比較しなければなりません。遺伝子発現および表現型が未処理の細胞とネガティブコントロールsiRNAをトランスフェクトした細胞において類似していることが理想です。ネガティブコントロールsiRNAをトランスフェクトした細胞で発現あるいは表現型の変化が観察された場合には、これらの変化はトランスフェクション操作あるいはsiRNAの毒性や配列の相補性がないことなどによる非特異的なものです。非特異的な効果は確実なRNAi/miRNA結果を確保するために最小限でなくてはなりません。
ネガティブコントロールでの結果は実験している遺伝子特異的なRNAi/miRNAによる結果と比較することもできます。ネガティブコントロール処理したサンプルはsiRNA/miRNA配列が違うだけで同じ生物学的な過程を経ているため、この両者を比較することにより研究者は標的遺伝子のノックダウンが遺伝子発現や表現型へ効果を及ぼしたかを正確に判断することができます。
AllStars Negative Control siRNAを用いた実験結果は以下のように利用できます:
Multiple negative control siRNAs (Control 1– Control 10) were transfected in triplicate into MCF-7 cells. After incubation, cRNA was prepared and hybridized to Affymetrix human U133 GeneChip arrays. Regulated genes were identified as genes that showed at least a 1.5-fold change in expression (both upregulated and downregulated) compared to untransfected cells. Ingenuity pathway analysis software was used to determine the proportion of regulated genes in each pathway compared to the total number of genes identified as central to that pathway. Where a bar appears in the figure, this means that genes in the pathway were regulated by the siRNA. If every pathway gene was regulated, the relative proportion would be 100%. Lower bars therefore indicate a lower relative proportion of regulated genes within that pathway. Where no bar appears, no genes of the pathway were regulated by the siRNA. AllStars Negative Control siRNA (indicated with arrow) resulted in the lowest number of regulated genes. In contrast, other control siRNAs resulted in higher numbers of regulated genes from important cellular pathways.
| Features | Specifications |
|---|---|
| Design | Predesigned/validated by Affymetrix GeneChip Array and cell-based assays |
| Species | Human, mouse, rat |
| Scale or yield | 5 nmol, 20 nmol |
| Format | Tube |
| Target sequence provided | No |
| Modification | Yes |