✓ 24/7 automatic processing of online orders
✓ Knowledgeable and professional Product & Technical Support
✓ Fast and reliable (re)-ordering
Cat. No. / ID: 206343
✓ 24/7 automatic processing of online orders
✓ Knowledgeable and professional Product & Technical Support
✓ Fast and reliable (re)-ordering
Features
- 複数の変異を高い再現性で正確に解析
- 至適化実験なしにマルチプレックスPCRアッセイを確立
- 全フラグメントを特異的かつ高感度で同時増幅
- 至適化済みプロトコールで正確な結果を迅速に実現
- 各種解析用装置を使用する場合の簡単なインストラクション
Product Details
Type-it Mutation Detect PCR Kitは、欠失、挿入、転座のような変異を迅速かつ確実に検出するために特別にデザインされています。特異性の高いHotStarTaq Plus DNA Polymerase と特許を持つバッファーシステムをベースにした本キットは、マルチプレックスPCR により遺伝子座の変異を確実に増幅し、至適化は不要です。続く解析は、アガロースゲル、自動化電気泳動装置、高分離能キャピラリーシークエンシングでも可能で迅速かつ容易に行なえます。
Performance
See figures
Principle
Type-it Mutation Detect PCR Kit は、実証済みのQIAGENマルチプレックステクノロジーをベースとし、即使用可能なマスターミックスフォーマットで提供されます。本マスターミックスは至適化済みの濃度のHotStarTaq Plus DNA Polymerase、MgCl2、dNTPsと、さらにマルチプレックスPCRベースの変異検出やゲノムSNP遺伝子座の事前増幅用に特別に開発された画期的なPCRバッファーが入っています。また、塩類とバランスよく配合された画期的な添加物であるFactor MP が入っています。これらの成分により、すべてのプライマーのアニーリングおよびエクステンションにおいて同等の効率化を可能にし(図“ 安定で効果的なアニーリング”)、同時に全フラグメントの高収量なマルチプレックス増幅を高い信頼性で実現します。本キットには、変異検出のための専用プロトコール、テンプレート量、サイクル数、およびPCR条件に関するステップごとのアドバイスと同様に、アガロースゲル、キャピラリーシークエンサー、Agilent Bioanalyzer、QIAxcel Advanced Systemなどのダウンストリーム解析のプラットフォーム用ごとに機器の詳細が含まれています。
マスターミックスの成分と専用プロトコールにより、高い特異性ですべてのフラグメントを同時に増幅できます。続く解析は、アガロースゲル、自動化電気泳動装置、高分離能キャピラリーシークエンシングでも可能で迅速かつ容易に行なえます。
Type-it Mutation Detect PCR Buffer
Type-it Mutation Detect PCR Buffer は、多数のマルチプレックス実験においてすべてのアンプリコンを同等の効率で増幅できるため、より多くの標的変異を解析でき、ターゲットで増幅効率のロスがありません。Type-it Mutation Detect PCR Bufferは、特別に開発されたバランスの取れた塩と添加物を組み合わせて、従来のPCR試薬に比べ、反応中の全プライマーのアニーリングとエクステンションを同等の効率で増幅します。このバッファーはユニークな配合比のKClと (NH4)2SO4を含み、従来のPCRバッファーに比べ、幅広い温度やMg2+濃度の範囲で厳密で特異的なプライマーアニーリングを実現します。異なるアニーリング温度あるいはMg2+ 濃度を用いて行なうPCRの至適化は最小限ですみ、または不要なこともあります。従来のマルチプレックスPCR操作で行なう至適化操作は不要です。バッファーに入っている合成 Factor MP (図“ 安定で効果的なアニーリングの促進”)は、プライマー配列に関係なくプライマーのアニーリングとエクステンションを効率的に行ないます。Factor MPは、DNAテンプレート付近のプライマーの濃度を上げ、特異的に結合したプライマーを安定化します。
Q-Solution
Type-it Mutation Detect PCR Kit にはQ-Solutionが添付されていますこの革新的なPCR 添加剤はDNAの変性環境を改善することにより、増幅困難なテンプレートの増幅を可能にします。このユニークな試薬を使用すると、PCR条件が最適でない場合でもPCR が可能になり、改良されることがあります。DMSOや他のPCR 添加物とは異なり、Q-Solution はどのプライマー/テンプレートシステムでも一定のワーキング濃度で使用し、毒性はありません。
CoralLoad Dye
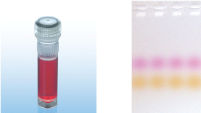
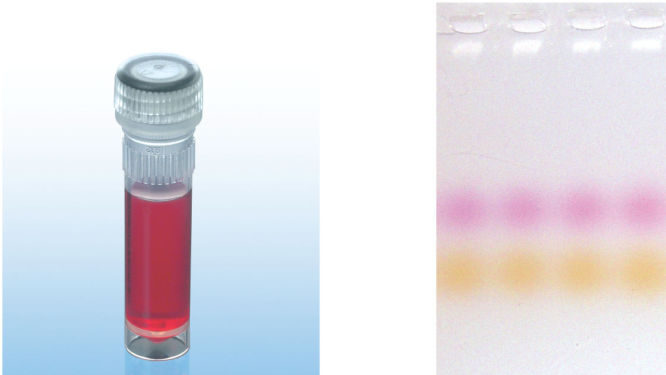
Type-it Mutation Detect PCR Kit に添付の CoralLoad Dye(図“ CoralLoad Dye”) には、ゲルローディング試薬と2種類のマーカー色素が入っており、DNAの移動距離の予測およびアガロースゲルの泳動時間の至適化が容易に行なえます。マルチプレックスPCR反応でCoralLoad Dye を使用する際は、増幅産物を直接アガロースゲルあるいはQIAxcel Advanced System にアプライします。ローディングバッファーを前もって添加する必要がありません。CoralLoad 色素はダウンストリームのほとんどの酵素アプリケーションで影響しません。しかし、再現性のある結果のためには、酵素処理を行なう前にPCR産物の精製を行なうことを推奨します。
検出にキャピラリーシークエンサーを使用する場合には、CoralLoad Dyeを使用しないでください。
See figures
Procedure
Type-it Mutation Detect PCR Kitは、アプリケーションに特化したプロトコールが入っており、それらはルーチン解析新しいアッセイの確立のため信頼性の高い結果を保証するため、同時増幅やその後の変異やSNP含む遺伝子座の検出用に最適化されています。反応は室温で設定することができるので、使いやすく便利です。
迅速かつ簡単な方法で信頼できるジェノタイピング結果を実現
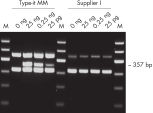
Type-it Mutation Detect PCR Kit は、マルチプレックス・アッセイを迅速かつ簡単に確立でき、正確で再現性のあるジェノタイピング結果が得られます。転座、欠失、挿入の検出、 SNaPshot (Applied Biosystems)のようなSNP検出前のゲノム遺伝子座の増幅などに、テンプレートとプライマーを添加し、至適化済みプロトコールに従ってサーマルサイクラープログラムをスタートするだけで実験ができます。反応ミックスには、変異に特異的なマルチプレックスPCRに必要な試薬が全て入っており、本キットを用いると、通常の方法のように時間がかかる至適化操作なしに正確な結果が得られます(図“ マルチプレックスPCRによる良好なジェノタイピング解析”)。
簡単な操作法により時間と経費を節約
いくつかの研究では、ある疾病に関与している特定遺伝子の様々な変異(例;欠失、転移、挿入)の多数解析が必要で、その場合singleplexあるいは2 ~ 3種類のマルチプレックスPCR 解析では膨大な数のPCR反応を実施しなければならず、その結果、経費および解析時間の増大に繋がります。Type-it Mutation Detect PCR Kitは、多数のマルチプレックス実験においてすべてのアンプリコンを同等の効率で増幅できるため、経費および実験時間を顕著に抑えてより多くの標的変異を解析できます (図 “ がん関連遺伝子の変異を高感度で検出”)。同時増幅やその後の変異やSNPを含む遺伝子座の検出のためのアプリケーションに特異的な専用プロトコールがキットに含まれ、ルーチン解析や新しいアッセイの確立のための信頼性の高い結果を保証します。
SNP 検出前の確実な増幅
Type-it Mutation Detect PCR Kit はまたSNP検出前のマルチプレックスPCRを利用した増幅にも最適です(例;Applied BiosystemsのSNaPshotシステムなど)。本キットは他社のキットより性能が優れ、至適化なしに正確な結果が一定して得られます(“ SNP 検出前の確実なマルチプレックス増幅”)。
See figures
Applications
Type-it Mutation Detect PCR Kitは欠失、挿入、重複、転座などの変異解析、またSNP検出前増幅(例;SNaPshot Multiplex Kit)のために使用でき、以下のような研究分野に利用できます。
- 疾患遺伝子座のタイピング
- GMO分析
- トランスジェネティック植物/動物のタイピング
Supporting data and figures
CoralLoad Dye.
Specifications
| Features | Specifications |
|---|---|
| Applications | Multiplex PCR, detection of mutations, preamplification of SNPs |
| Mastermix | Yes |
| Reaction type | PCR amplification |
| Product use | Functionally validated and developed for reliable mutation analyis |
| Real-time or endpoint | Endpoint |
| Sample/target type | Genomic DNA |
| Single or multiplex | Multiplex |
| With/without hotstart | With |